Yn it proses fan it útfieren fan genetyske stúdzjes komme wy faaks te min RNA-monsters tsjin, bygelyks foar it bestudearjen fan lytse anatomyske orale tumors, sels iensellige samples, en samples fan spesifike genmutaasjes dy't op heul lege nivo's yn minsklike sellen wurde transkribearre.Fansels, foar de COVID-19-test, as de swabs net op it goede plak binne of net genôch kearen tidens sampling, sil de stekproefgrutte heul leech wêze, dat is de reden dat de Kommisje foar sûnens en famyljeplanning twa dagen lyn kaam en slagge de test, en as de nucleic acid sampler net nimme seis samples, kinne jo melde it.
De gefoelichheid fan it reagens is wichtich om't wy dit probleem of dat probleem hawwe, dus wat kinne wy dwaan om de gefoelichheid fan RT-PCR te ferbetterjen?
Foardat wy mooglike oplossingen besprekke, litte wy twa grutte komplikaasjes neame mei de situaasje dy't wy krekt neamden.
Alderearst meitsje wy ús soargen oer RNA-ferlies as wy mar in pear selpopulaasjes yn ús stekproef hawwe.As tradisjonele metoaden foar skieding en skjinmeitsjen wurde brûkt, lykas kolommetoade of nukleïnesoere-ôfslachmetoade, is d'r in hege kâns dat de pear monsters ferlern gean.Ien oplossing is om in dragermolekule ta te foegjen, lykas tRNA, mar sels dan is d'r gjin garânsje dat ús hersteleksperimint OK is.
Dus wat is in bettere manier?In goede opsje foar kweekte sellen as mikroanatomyske samples is om direkte lysis te brûken.
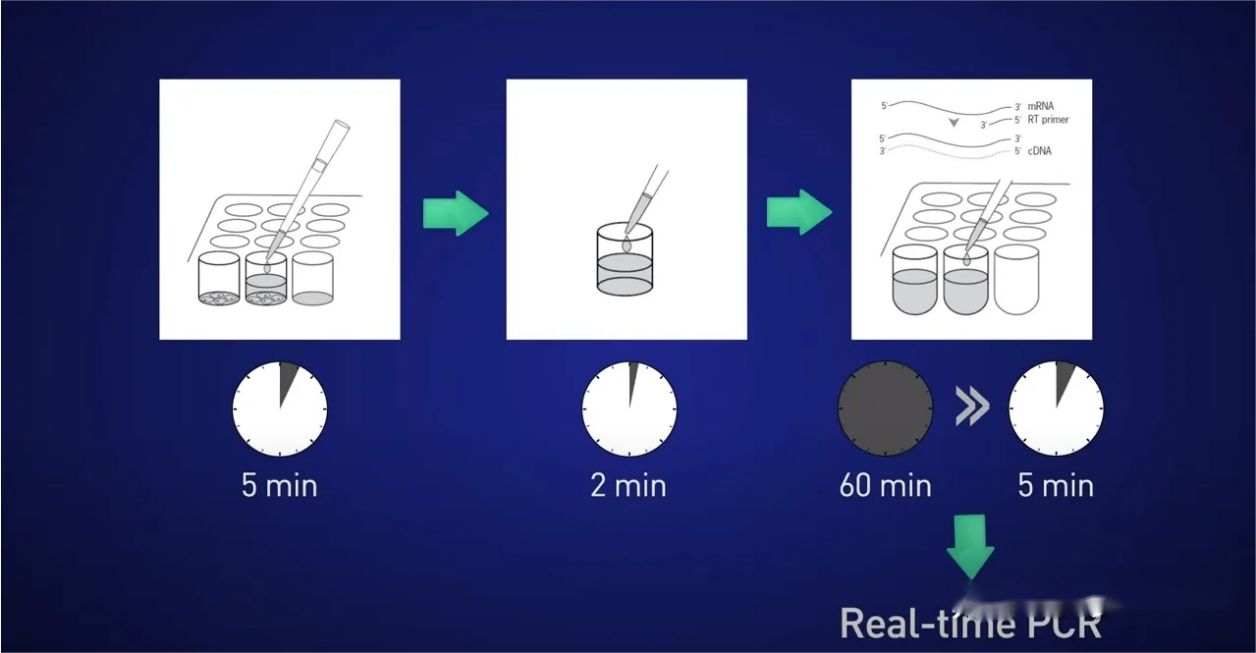
It idee is om de sellen foar 5 minuten te splitsen, it RNA frij te litten yn 'e oplossing, stopje dan de reaksje foar 2 minuten, foegje dan it lysate direkt ta oan 'e omkearde transkripsjereaksje sadat gjin RNA ferlern giet, en sette úteinlik de resultearjende cDNA direkt direkt yn 'e real-time reaksje.
Mar wat as wy, fanwege in beheind útgongspunt of in lyts bedrach fan doelgenekspresje, alle RNA kinne recycleje en noch net genôch sjabloanen leverje om in goed real-time sinjaal te krijen?
Yn dit gefal kin de pre-amplifikaasjestap heul nuttich wêze.
It folgjende is in skema om gefoelichheid te ferheegjen nei omkearde transkripsje.Foardat wy begjinne, moatte wy streamôfwerts freegje hokker doelen wy binne ynteressearre yn, om spesifike primers foar dizze doelen te ûntwerpen foar pre-amplifikaasje.
Dit kin berikt wurde troch it meitsjen fan in mingde primer mei maksimaal 100 pearen primers en in reaksjesyklus fan 10 oant 14 kear.Dêrom is in Master Mix spesifyk ûntworpen foar dizze eask nedich om it krigen cDNA foarôf te fersterkjen.
De reden foar it ynstellen fan it oantal syklusen tusken 10 en 14 is dat dit beheinde oantal syklusen soarget foar willekeurigens tusken de ferskate doelen, wat krúsjaal is foar ûndersikers dy't kwantitative molekulêre ynformaasje nedich binne.
Nei pre-amplifikaasje kinne wy in grutte hoemannichte cDNA krije, sadat de deteksjegefoelichheid oan 'e efterkant sterk ferbettere wurdt, en wy kinne sels de stekproef ferwetterje en meardere real-time PCR-reaksjes útfiere om mooglike willekeurige flaters te eliminearjen.
Post tiid: Apr-11-2023